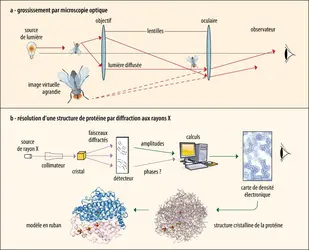
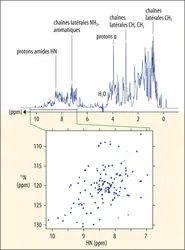
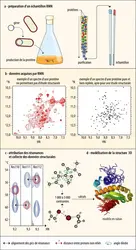
DÉTERMINATION DE LA STRUCTURE 3D DES PROTÉINES
Article modifié le
L’apport de l’intelligence artificielle
En 1972, lorsqu’il reçut le prix Nobel de chimie à Stockholm, Christian Anfinsen déclarait : « Des considérations empiriques sur la grande quantité de données actuellement disponibles qui corrèlent la séquence et la structure tridimensionnelle, ainsi qu'une sophistication croissante dans le traitement théorique des aspects énergétiques du repliement des chaînes polypeptidiques, commencent à rendre plus réaliste l'idée d'une prédiction a priori de la conformation des protéines. »
Ainsi, depuis longtemps, des scientifiques réfléchissent et travaillent sur la prédiction des structures de protéines à partir de leurs séquences. En 2021, un algorithme, nommé AlphaFold et développé par DeepMind, a surpassé tous les autres algorithmes de prédiction de structures 3D des protéines ; il révolutionne, dès lors, la biologie structurale. Il s’agit d’un logiciel d’apprentissage automatique qui a été initialement entraîné à partir des structures de protéines obtenues auparavant par expérimentation. En peu de temps, l’utilisation d’AlphaFold a produit un nombre gigantesque de structures théoriques de protéines : « AlphaFold offre un accès libre à plus de 200 millions de prédictions de structures protéiques [en 2024] afin d'accélérer la recherche scientifique », surpassant le nombre de structures expérimentales d’un facteur 1 000. Il est important de rappeler que cet algorithme est fondé sur les connaissances acquises par la biologie structurale expérimentale et donc sur les milliers de structures déjà déterminées expérimentalement. Il fournit des modèles prédits et ne peut donc pas entièrement se subsister aux techniques expérimentales qui, en revanche, peuvent les vérifier et les utiliser.
L'essor de l’intelligence artificielle bénéficie dans une certaine mesure à la biologie structurale. Comme précédemment mentionné, les techniques expérimentales utilisent déjà l'information structurale produite par AlphaFold : posséder un modèle structural relativement fiable, issu d’une prédiction, peut significativement accélérer les processus complexes de la détermination structurale expérimentale. Pour la cristallographie aux RX, l’apport d’AlphaFold se concentre dans la génération de modèles assez précis pour appliquer la technique de remplacement moléculaire à la résolution des structures pour lesquelles soit nous ne disposons pas de modèles expérimentaux semblables, soit ces derniers ne sont pas assez proches de la protéine étudiée. Pour la RMN, des modèles structuraux générés par l'intelligence artificielle ouvrent à une interprétation plus fiable (car moins ambiguë) des données et donc la détermination de structures de meilleure qualité. Aussi, ces modèles peuvent être utiles pour l'interprétation d'autres données RMN liées à la fonction d'une protéine (dynamique, interactions...). Pour la cryomicroscopie, AlphaFold permet de pallier une résolution moindre (au-delà de 4-5 Å) obtenue dans certains cas, comme dans celui de complexes multiprotéiques, et joue alors un rôle de complémentarité avec les données de cryo-ME. En effet, à partir des séquences de chaque protéine individuelle constituant le complexe, AlphaFold va pouvoir respectivement prédire leur structure 3D. Ces structures vont alors être positionnées à l'intérieur des cartes de cryo-ME et ainsi permettre de décrire des interactions au niveau atomique entre les différents constituants du complexe.
Alors que la prédiction de la structure d'une protéine est une affaire de secondes, les expérimentalistes travaillent à utiliser ces modèles sans pour autant abandonner les méthodes expérimentales, qui restent indispensables et complémentaires pour valider les modèles obtenus par prédiction.
La suite de cet article est accessible aux abonnés
- Des contenus variés, complets et fiables
- Accessible sur tous les écrans
- Pas de publicité
Déjà abonné ? Se connecter
Écrit par
- Beate BERSCH : chargée de recherche au CNRS
- Juan FONTECILLA-CAMPS : conseiller scientifique au CEA
- Emmanuelle NEUMANN : docteure en physique, ingénieure-chercheuse au CEA Grenoble
Classification
Médias
Voir aussi
- FRÉQUENCE, physique
- CONGÉLATION
- NOYAU ATOMIQUE
- MICROSCOPIE ÉLECTRONIQUE
- MICROSCOPIE ÉLECTRONIQUE À BALAYAGE EN TRANSMISSION
- INTERACTIONS MOLÉCULAIRES
- STRUCTURE, biologie
- RMN (résonance magnétique nucléaire), physico-chimie
- CRISTALLOGRAPHIE
- DIFFRACTION PAR LES CRISTAUX
- BIOLOGIE MOLÉCULAIRE
- HÉLICE, chimie des protéines
- LARMOR FRÉQUENCE DE
- MICROSCOPIE ÉLECTRONIQUE À BALAYAGE
- MOLÉCULES BIOLOGIQUES, structure et fonction
- MACROMOLÉCULES BIOLOGIQUES
- ISOTOPES, biologie
- TOMOGRAPHIE
- PROTÉINES
- BIOLOGIE HISTOIRE DE LA
- IMAGE ou REPRÉSENTATION TRIDIMENSIONNELLE (3D)