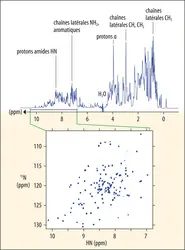
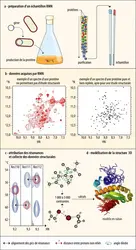
DÉTERMINATION DE LA STRUCTURE 3D DES PROTÉINES
Article modifié le
Sites internet
AlphaFold Protein Structure Database, https://alphafold.ebi.ac.uk/
Worldwide Protein Data Bank, https://www.wwpdb.org/
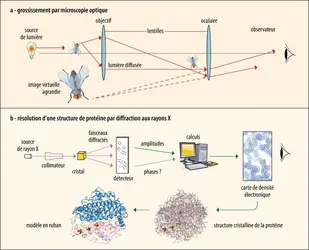
Cristallographie aux rayons X
Association française de cristallographie, https://www.afc.asso.fr/
Cornell High Energy Synchrotron Source (CHESS), Serial Crystallography, https://www.chess.cornell.edu/macchess/mx/sx
International Year of Cristallography 2014, https://www.iycr2014.org/learn/crystallography365/articles/20140828
Mettler Toledo, Cristallisation des proteines, https://www.mt.com/fr/fr/home/applications/L1_AutoChem_Applications/L2_Crystallization/protein-crystallization.html
UNS Nice, Bandes interdites phototoniques, http://physique.unice.fr/sem6/2009-2010/PagesWeb/Cristaux/bande%20interdites.html
Cryomicroscopie électronique
Electron Microscopy Data Bank (EMDB), https://www.ebi.ac.uk/emdb/statistics ; « EMD-19436. Cryo-EM structure of mouse heavy-chain apoferritin », https://www.ebi.ac.uk/emdb/EMD-19436
La suite de cet article est accessible aux abonnés
- Des contenus variés, complets et fiables
- Accessible sur tous les écrans
- Pas de publicité
Déjà abonné ? Se connecter
Écrit par
- Juan FONTECILLA-CAMPS : conseiller scientifique au CEA
- Beate BERSCH : chargée de recherche au CNRS
- Emmanuelle NEUMANN : docteure en physique, ingénieure-chercheuse au CEA Grenoble
Classification
Médias