GÉNOMIQUE : ANNOTATION DES GÉNOMES
Article modifié le
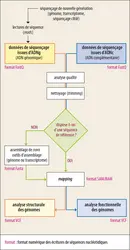
À l’exception de certains virus, l’information génétique nécessaire au métabolisme et à la perpétuation des organismes est contenue dans l’ADN de leurs chromosomes, dans les gènes de leurs génomes et dans les régions régulatrices qui contrôlent leur activité. Cette information est codée dans un alphabet à quatre lettres, les quatre bases organiques des nucléotides – adénine, thymine, cytosine et guanine (ATCG) –, au long de la molécule d’ADN de chaque chromosome. Connaître la séquence nucléotidique d’un génome, c’est avoir accès à ses gènes, à ce qui détermine les caractères physiques d’un organisme – son phénotype – et son adaptabilité aux variations de son environnement. En d’autres termes, déterminer la séquence d’un génome permettrait de « tout connaître » d’un organisme. L’affirmation est quelque peu naïve, mais il n’en reste pas moins que disposer de la séquence d’un génome est devenu une étape essentielle dans la connaissance d’un organisme, ce qui n’est pas pour autant facile à réaliser : des séquences génomiques fiables sont obtenues au prix de consolidations successives des séquences qui sortent du séquenceur d’ADN. C’est ce dont il sera surtout question ici.
Construire la « bibliothèque de Babel » des génomes
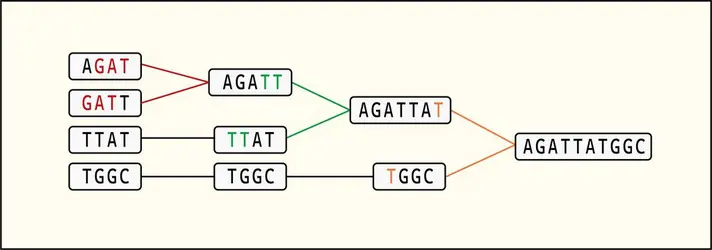
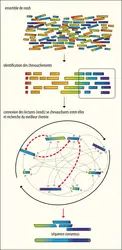
Depuis le lancement du projet de séquençage du génome humain en octobre 1990, les techniques de séquençage de l’ADN génomique ont évolué de manière spectaculaire. La méthode historique, dite par terminaison de chaîne, mise au point par Frederick Sanger (1918-2013) en 1977, permettait, en une seule réaction, de déterminer la séquence d’un fragment d’ADN sur une longueur de 1 000 nucléotides au maximum. L’automatisation de cette technique, à partir des années 1990 par les séquenceurs dits de première génération, a contribué à l’obtention de nombreux génomes complets : premier génome bactérien en 1995 (Haemophilus influenzae), premier génome eucaryote en 1996 (Saccharomyces cerevisiae), suivis de ceux des principaux organismes modèles jusqu’à la souris Mus musculus en 2002, sans oublier bien sûr celui d’Homo sapiens en 2001 dans sa version « brouillon », au prix d’un effort de dix ans et d’un coût de 3 milliards de dollars pour une longueur d’environ 3 milliards de nucléotides. Ces quinze années de recherche (1990-2005) ont constitué la première révolution génomique.
À partir de 2005, l’ère de la deuxième révolution génomique commence, avec le développement des approches de séquençage dites de nouvelle génération (NGS, New Generation Sequencing). Les séquenceurs NGS permettent de séquencer plusieurs millions – voire 1 milliard – de fragments différents en parallèle. Cette pratique a effondré le coût du séquençage, qui passe, pour l’exemple du génome humain, à 1 000 dollars en 2015 – la séquence est obtenue en quelques jours –, démocratisant ainsi son utilisation du point de vue financier. De nombreux laboratoires de recherche, même les plus modestes, ont désormais accès au génome des organismes qu’ils étudient. On peut en effet désormais « tout » séquencer à des coûts abordables : ADN génomique, ARN messager (ARNm, préalablement rétrotranscrit en ADNc), ADN ou ARN ciblés, grâce à la mise en place d’un protocole d’enrichissement (analyse de l’exome, de l’épigénome, etc.). Tout génome étant devenu accessible, le dernier en date des grands programmes de séquençage, Earth BioGenome Project, lancé en janvier 2022, envisage de séquencer le génome d’au moins un spécimen chez chacune des 1 800 000 espèces vivantes connues, bref créer une sorte de bibliothèque de Babel des génomes.
Il s’agit désormais de savoir interpréter cette masse d’informations, essentiellement accessible à tous, pour répondre à une multitude de questions, de la variation d’un virus aux[...]
La suite de cet article est accessible aux abonnés
- Des contenus variés, complets et fiables
- Accessible sur tous les écrans
- Pas de publicité
Déjà abonné ? Se connecter
Écrit par
- Véronique BLANQUET : professeure de génétique, université de Limoges
- Stéphanie DURAND : maître de conférences, faculté des sciences et techniques de Limoges
Classification
Médias
Autres références
-
SÉQUENÇAGE DU GÉNOME HUMAIN, en bref
- Écrit par Nicolas CHEVASSUS-au-LOUIS et Encyclopædia Universalis
- 286 mots
Le 12 février 2001, les revues scientifiques Nature et Science publient la séquence quasi complète des trois milliards de bases du génome humain. Cette double publication conclut par un ex aequo la compétition entre un consortium international de laboratoires publics, qui a commencé ses...
-
BIOTECHNOLOGIES
- Écrit par Pierre TAMBOURIN
- 5 368 mots
- 4 médias
Des gènes responsables de la résistance aux herbicides, aux infections virales, fongiques ou bactériennes, identifiés grâce au progrès de lagénomique, sont utilisés pour transférer aux plantes ces mêmes propriétés de résistance. C'est en 1985 que les premiers essais en champ de plantes transgéniques... -
BOTANIQUE
- Écrit par Sophie NADOT et Hervé SAUQUET
- 5 647 mots
- 7 médias
...recherche scientifique s'est accélérée, engendrant des bouleversements imprévisibles de notre compréhension du monde, y compris celui des plantes. La génomique, par exemple, est en train de révéler une évolution et un fonctionnement bien plus complexe des génomes d'eucaryotes (organismes pourvus d'un... -
CANCER - Cancer et santé publique
- Écrit par Maurice TUBIANA
- 14 762 mots
- 8 médias
... représente un autre domaine de recherche. Maintenant qu'ont été identifiés les défauts du génome caractérisant les cellules cancéreuses, il est tentant d'essayer de les corriger et de faire redevenir normales les cellules cancéreuses. Quelques résultats ont été obtenus chez l'homme dans... - Afficher les 29 références
Voir aussi
- NUCLÉOTIDES
- GÉNOTYPE
- CODON, biologie moléculaire
- ARN MESSAGER ou ARNm
- EUCARYOTES
- SÉQUENÇAGE, génétique moléculaire
- GÉNOME
- THÉRAPIE GÉNIQUE
- BIOLOGIE MOLÉCULAIRE
- BANQUE DE DONNÉES
- CHROMOSOMES
- EXPRESSION GÉNÉTIQUE
- EXON
- INTRON
- NUCLÉOTIDIQUE SÉQUENCE
- GÉNÉTIQUE MOLÉCULAIRE
- TECHNIQUES HISTOIRE DES, XXe et XXIe s.
- PROTÉINES
- ADN COMPLÉMENTAIRE ou ADNc