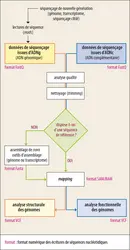
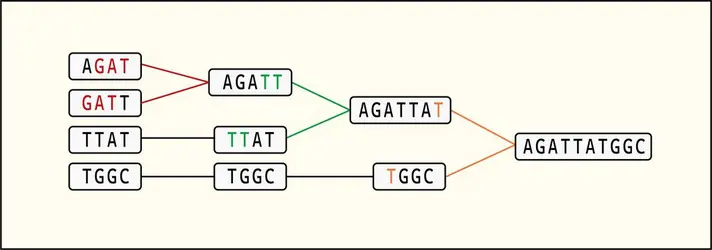
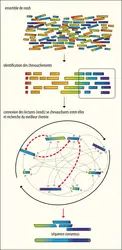
GÉNOMIQUE : ANNOTATION DES GÉNOMES
Article modifié le
Bibliographie
J. Commins, C. Toft & M. A. Fares, « Computational biology methods and their application to the comparative genomics of endocellular symbiotic bacteria of insects », in BiologicalProcedures Online, vol. 11, pp. 52-78, 2009 (https://doi.org/10.1007/s12575-009-9004-1)
K. Eilbeck, A. Quinlan & M. Yandell, « Settling the score: variant prioritization and Mendelian disease », in Nature Reviews, Genetics, vol. 18, no 10, pp. 599-612, 2017
N. A. Fonseca, J. Rung, A. Brazma & J. C. Marioni, « Tools for mapping high-throughput sequencing data », in Bioinformatics, vol. 28, no 24, pp. 3169-3177, 2012 (https://doi.org/10.1093/bioinformatics/bts605)
GenomeAsia100K Consortium, The GenomeAsia 100K Project enables genetic discoveries across Asia, in Nature, vol. 576, pp. 106-111, 2019
S. Goodwin, J. McPherson & W. McCombie, « Coming of age: ten years of next-generation sequencing technologies », in Nature Reviews, Genetics, vol. 17, no 6, pp. 333-351, 2016
S. Gudmunsson, M. Singer-Berk, N. A. Watts et al., « Variant interpretation using population databases: Lessons from gnomAD », in Human Mutation, vol. 43, no 8, pp. 1012-1030, 2021 (https://doi.org/10.1002/humu.24309)
D. C. Koboldt, « Best practices for variant calling in clinical sequencing », in Genome Medicine, vol. 12, no 1, pp. 91-114, 2020 (https://genomemedicine.biomedcentral.com/articles/10.1186/s13073-020-00791-w)
H. Li, B. Handsaker, A. Wysoker et al., « The sequence alignment/map format and SAMtools, in Bioinformatics, vol. 25, no 16, pp. 2078-2079, 2009 (https://doi.org/10.1093/bioinformatics/btp352)
S. P. Pfeifer, « From next-generation resequencing reads to a high-quality variant data set», in Nature Heredity, vol. 118, no 2, pp. 111-124, 2017 (https://www.nature.com/articles/hdy2016102)
F. Sanger, S. Nicklen & A. R. Coulson, « DNA sequencing with chain-terminating inhibitors », in Proceedings of the National Academy of Sciences of the USA, vol. 74, no 12, pp. 5463-5467, 1977 (https://doi.org/10.1073/pnas.74.12.5463)
Z. Wang, Y. Chen & Y. Li, « A brief review of computational gene prediction methods », in Genomics Proteomics Bioinformatics, vol. 2, no 4, pp. 216-221, 2004 (https://doi.org/10.1016/s1672-0229(04)02028-5)
1,000 Genomes Project Consortium, « A global reference for human genetic variation », in Nature, vol. 526, pp. 68–74, 2015 (https://www.nature.com/articles/nature15393)
100,000 Genomes Project Pilot Investigators, « Genomes Pilot on Rare-Disease Diagnosis in Health Care – Preliminary Report », in The New England Journal of Medicine, vol. 385, no 20, pp. 1868-1880, 2021 (https://doi.org/10.1056/NEJMoa2035790).
Sites Internet
FASTQC : https://www.bioinformatics.babraham.ac.uk/projects/fastqc/Help/
GATK (Genome Analysis ToolKit) : https://gatk.broadinstitute.org/hc/en-us
Projet POPGEN, Diversité génomique de la population française : https://lysine.univ-brest.fr/popgen/
La suite de cet article est accessible aux abonnés
- Des contenus variés, complets et fiables
- Accessible sur tous les écrans
- Pas de publicité
Déjà abonné ? Se connecter
Écrit par
- Véronique BLANQUET : professeure de génétique, université de Limoges
- Stéphanie DURAND : maître de conférences, faculté des sciences et techniques de Limoges
Classification
Médias
Autres références
-
SÉQUENÇAGE DU GÉNOME HUMAIN, en bref
- Écrit par Nicolas CHEVASSUS-au-LOUIS et Encyclopædia Universalis
- 286 mots
Le 12 février 2001, les revues scientifiques Nature et Science publient la séquence quasi complète des trois milliards de bases du génome humain. Cette double publication conclut par un ex aequo la compétition entre un consortium international de laboratoires publics, qui a commencé ses...
-
BIOTECHNOLOGIES
- Écrit par Pierre TAMBOURIN
- 5 368 mots
- 4 médias
Des gènes responsables de la résistance aux herbicides, aux infections virales, fongiques ou bactériennes, identifiés grâce au progrès de lagénomique, sont utilisés pour transférer aux plantes ces mêmes propriétés de résistance. C'est en 1985 que les premiers essais en champ de plantes transgéniques... -
BOTANIQUE
- Écrit par Sophie NADOT et Hervé SAUQUET
- 5 647 mots
- 7 médias
...recherche scientifique s'est accélérée, engendrant des bouleversements imprévisibles de notre compréhension du monde, y compris celui des plantes. La génomique, par exemple, est en train de révéler une évolution et un fonctionnement bien plus complexe des génomes d'eucaryotes (organismes pourvus d'un... -
CANCER - Cancer et santé publique
- Écrit par Maurice TUBIANA
- 14 762 mots
- 8 médias
... représente un autre domaine de recherche. Maintenant qu'ont été identifiés les défauts du génome caractérisant les cellules cancéreuses, il est tentant d'essayer de les corriger et de faire redevenir normales les cellules cancéreuses. Quelques résultats ont été obtenus chez l'homme dans... - Afficher les 29 références
Voir aussi
- NUCLÉOTIDES
- GÉNOTYPE
- CODON, biologie moléculaire
- ARN MESSAGER ou ARNm
- EUCARYOTES
- SÉQUENÇAGE, génétique moléculaire
- GÉNOME
- THÉRAPIE GÉNIQUE
- BIOLOGIE MOLÉCULAIRE
- BANQUE DE DONNÉES
- CHROMOSOMES
- EXPRESSION GÉNÉTIQUE
- EXON
- INTRON
- NUCLÉOTIDIQUE SÉQUENCE
- GÉNÉTIQUE MOLÉCULAIRE
- TECHNIQUES HISTOIRE DES, XXe et XXIe s.
- PROTÉINES
- ADN COMPLÉMENTAIRE ou ADNc