MULLIS KARY BANKS (1944-2019)
Kary Banks Mullis est un biologiste moléculaire américain né le 28 décembre 1944 à Lenoir (Caroline du Nord). Il fait ses études à l'institut de technologie de Georgie et à l'université de Berkeley (Californie) où il obtient son doctorat en 1973. Après avoir effectué des recherches à l'école médicale de l'université du Kansas puis à l'université de Californie à San Francisco, il rejoint en 1979 une entreprise de biotechnologie, la Cetus Corporation, à Emeryville (Californie), où il travaille à la synthèse d'oligonucléotides. Il exerce ensuite comme consultant indépendant et travaille à diverses inventions. Kary Mullis meurt le 7 août 2019 à Newport Beach (Californie).
Comme il l'a lui-même raconté dans son autobiographie (Dancing Naked in the Mind Field, 1998), c'est durant une nuit de 1983, alors qu'il effectuait un long parcours automobile, que Mullis eut l'idée de la découverte qui allait faire sa renommée et lui permettre de partager avec Michael Smith le prix Nobel de chimie en 1993. Le problème qui le préoccupait était de trouver une méthode permettant d'identifier des gènes ou d'autres fragments d'un acide désoxyribonucléique (ADN), à partir d'une très petite quantité de substance.
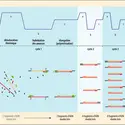
La méthode PCR (polymerasechainreaction), ou réaction de polymérisation en chaîne, mise au point par Mullis débute par la dénaturation thermique (95 0C) de l'échantillon d'ADN que l'on désire amplifier. La double hélice de l'ADN est alors séparée en ses deux brins. Des oligonucléotides, qui vont jouer le rôle d'amorces spécifiques complémentaires à l'ADN, sont préparés par synthèse chimique, ajoutés en excès à l'ADN dénaturé et incubés à 60 0C. Les amorces spécifiques s'accrochent en bonne position sur chacun des brins. À partir de ces points d'ancrage, une enzyme cellulaire, une ADN polymérase, va reconstituer un nouveau brin d'ADN en regard du brin original d'ADN grâce aux oligonucléotides, toujours présents dans le milieu réactionnel, qui permettent l'appariement spécifique des bases complémentaires dans la phase d'élongation. Le cycle de duplication, dont la durée est de quelques minutes, peut être répété pour amplifier la séquence d'ADN concernée, et, en quelques heures, 100 milliards de copies de chaque molécule initiale pourront être obtenues : l'ADN d'un seul bulbe pileux, soit un milliardième de gramme, peut ainsi être amplifié rapidement. La méthode PCR a vite trouvé de très nombreuses applications : cartographie et analyse de génomes, outil de diagnostic pour des maladies causées par des défauts de l'ADN (maladies génétiques, cancers), détection des virus (VIH) et des bactéries, investigation policière, médecine légale, etc.
La suite de cet article est accessible aux abonnés
- Des contenus variés, complets et fiables
- Accessible sur tous les écrans
- Pas de publicité
Déjà abonné ? Se connecter
Écrit par
- Georges BRAM : professeur à l'université de Paris-Sud-XI-Orsay
Classification
Média
Autres références
-
PCR (polymerase chain reaction) ou AMPLIFICATION EN CHAÎNE PAR POLYMÉRASE
- Écrit par Véronique BARRIEL
- 5 112 mots
- 6 médias
...qui vivent dans des conditions extrêmes – ici dans des sources chaudes – et dont l'ADN polymérase fonctionnant à haute température a été sélectionnée. L' assemblage de ces connaissances issues de divers domaines de la chimie de l’ADN en une technique homogène – la PCR – est dû au groupe dirigé par... -
POLYMERASE CHAIN REACTION (PCR)
- Écrit par Nicolas CHEVASSUS-au-LOUIS et Encyclopædia Universalis
- 352 mots