PCR (polymerase chain reaction) ou AMPLIFICATION EN CHAÎNE PAR POLYMÉRASE
Article modifié le
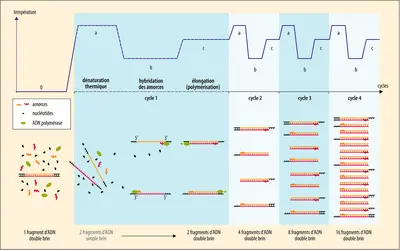
La PCR (polymerasechainreaction ou « amplification en chaîne par polymérase », expression française rarement utilisée) est une suite de réactions enzymatiques qui permettent d'amplifier un fragment d'ADN spécifique (ADN cible), souvent présent au départ en très faible quantité, et parfois en mauvais état, parmi des millions d'autres fragments. On peut mesurer la puissance de cette technique en soulignant sa capacité à produire, en quelques heures, plusieurs centaines de microgrammes de l'ADN d'un gène à partir de moins de 1 picogramme de ce même gène, soit une amplification de l'ordre du milliard de fois. Elle permet donc de s'affranchir des opérations longues, complexes et aléatoires qui étaient nécessaires au tout début de la biologie moléculaire pour isoler l'ADN d'un gène et en obtenir des quantités manipulables. La PCR est ainsi souvent considérée comme un véritable « photocopieur » de séquences d'ADN.
Rapidement, les applications de la PCR, en biologie et dans d’autres domaines, se sont multipliées. On utilise la PCR dans presque toutes les activités de base de la biologie moderne, mais également dans des applications dérivées, comme la détection de micro-organismes pathogènes, le diagnostic prénatal des maladies génétiques, le typage tissulaire, l'identification d'individus, la détermination de la filiation, le suivi des espèces protégées… – exemples qui appartiennent à des domaines aussi différents que la médecine, l'évolution, l'archéologie, l'industrie agroalimentaire, la justice, les biotechnologies, l'environnement... La PCR est ainsi devenue un outil polyvalent, une sorte de « couteau suisse » des biologistes au sens le plus large, utilisé pour aborder des problèmes jusque-là inaccessibles.
La PCR, un bricolage moléculaire
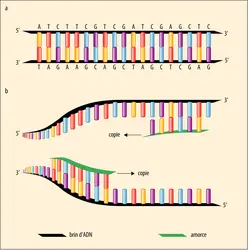
La PCR repose sur une sorte de « bricolage », qui associe diverses connaissances sur l'ADN. Au début des années 1950, on montre que l'ADN est une double hélice dont chaque brin s'enroule autour de l'autre selon la complémentarité de la séquence de leurs bases nucléiques : en face d'un A se trouve un T, en face d'un C se trouve un G. Il est possible de dissocier les brins par chauffage, et cela de manière réversible ; ils se réassocient correctement au cours du refroidissement. La notion clé de la PCR est la manière dont deux brins d'ADN se reconnaissent et s'apparient. Dès qu'un fragment d'ADN dépasse une certaine longueur (environ une vingtaine de bases), ce fragment peut s'associer très précisément à la région de séquence complémentaire d'un brin d'ADN. La PCR est fondée sur ce principe de complémentarité des séquences de bases et sur celui de la dissociation-réassociation sous l'effet de changements de température. Au début des années 1960, on montre que la synthèse de chaque brin est assurée par des enzymes, les ADN polymérases, qui fabriquent un brin neuf d'ADN par copie de l'autre brin, en partant d'une extrémité. Dans la PCR, on apparie, à l'extrémité de chaque brin de l'ADN d'un gène, un court fragment d'ADN d'une vingtaine de bases (appelé amorce) dont la séquence est complémentaire de celle qui est présente à chaque extrémité de la séquence d'ADN du gène ; ensuite, une polymérase complète la séquence d'ADN à partir de ces amorces en copiant le brin auquel l'amorce s'est fixée. Si l’on veut répéter l'opération, il faut dissocier les brins et les amorces entre chaque synthèse pour permettre le retour aux conditions initiales d'appariement. Cela se fait par chauffage. Mais, comme le chauffage détruit la plupart des polymérases, il faut en ajouter pour recommencer le cycle de synthèse. La solution à ce dernier problème est venue de l'étude d'organismes qui vivent dans des conditions extrêmes – ici dans des sources chaudes – et dont l'ADN polymérase fonctionnant à haute température a été sélectionnée. L' assemblage de ces connaissances[...]
La suite de cet article est accessible aux abonnés
- Des contenus variés, complets et fiables
- Accessible sur tous les écrans
- Pas de publicité
Déjà abonné ? Se connecter
Écrit par
- Véronique BARRIEL : maître de conférences au Muséum national d'histoire naturelle, Paris
Classification
Médias
Autres références
-
POLYMERASE CHAIN REACTION (PCR)
- Écrit par Nicolas CHEVASSUS-au-LOUIS et Encyclopædia Universalis
- 352 mots
-
BACTÉRIOLOGIE
- Écrit par Jean-Michel ALONSO , Jacques BEJOT , Michel DESMAZEAUD , Didier LAVERGNE et Daniel MAZIGH
- 18 335 mots
- 11 médias
...une sensibilité permettant de détecter parfois jusqu'à une seule copie d'une séquence génomique spécifique. La méthode la plus connue est la réaction de polymérisation en chaîne, ou P.C.R. (pour polymerase chain reaction). Elle utilise la propriété de l'enzyme ADN polymérase de synthétiser le brin complémentaire... -
BIOLOGIE - La biologie moléculaire
- Écrit par Gabriel GACHELIN
- 7 405 mots
- 8 médias
...définies de plus en plus longues et maintenant d'ARN, ce qui permet nombre de développements techniques. Enfin, une technique majeure introduite en 1985, la polymerase chain reaction (PCR), permet l'amplification d'à peu près n'importe quelle séquence d'ADN à partir de quantités aussi faibles que l'ADN... -
DENGUE
- Écrit par Philippe DESPRÈS
- 2 880 mots
- 4 médias
Pendant la phase aiguë de la maladie, la virémie est recherchée parPCR en temps réel, laquelle utilise des couples d'amorces capables de reconnaître toutes les souches virales ou, au contraire, spécifiques de chacun des sérotypes de la dengue. L'isolement viral à partir d'un sérum virémique est réalisé... -
DIAGNOSTIC VIROLOGIQUE
- Écrit par Yannick SIMONIN
- 5 194 mots
- 4 médias
...virales et cellulaires, ce qui permet d’extraire le génome viral normalement protégé à l’intérieur des capsides virales ou au sein des cellules infectées. L’amplification génique peut ensuite être effectuée. Elle repose sur la technique de polymérisation en chaîne (PCR pour polymerase chain reaction). - Afficher les 23 références
Voir aussi
- IDENTITÉ JUDICIAIRE
- ENZYMES
- MICRO-ORGANISME
- HYBRIDATION MOLÉCULAIRE
- HÉRÉDITAIRES MALADIES ou MALADIES GÉNÉTIQUES
- ADN POLYMÉRASE
- ARN MESSAGER ou ARNm
- DIAGNOSTIC PRÉIMPLANTATOIRE
- INFECTION
- SÉQUENÇAGE, génétique moléculaire
- GÉNOME
- BIOLOGIE MOLÉCULAIRE
- AGROALIMENTAIRE TECHNOLOGIE
- DIAGNOSTIC PRÉNATAL
- CYTOLOGIE ou BIOLOGIE CELLULAIRE
- AMPLIFICATION GÉNIQUE IN VITRO
- EXPRESSION GÉNÉTIQUE
- EMPREINTES GÉNÉTIQUES
- NUCLÉOTIDIQUE SÉQUENCE
- RECONNAISSANCE, biologie
- GÉNÉTIQUE MOLÉCULAIRE
- TRANSCRIPTASE INVERSE ou REVERSE TRANSCRIPTASE
- PHYLOGÉNIE MOLÉCULAIRE
- PALÉOGÉNÉTIQUE ET PALÉOGÉNOMIQUE
- ADN ANCIEN
- TAQ POLYMÉRASE