PRIX LASKER 2023
Article modifié le
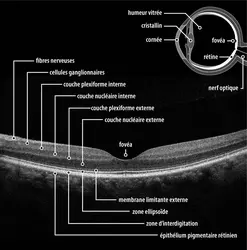
Les prix 2023 de la fondation Albert-et-Mary-Lasker, plus communément appelés « prix Lasker » et tenus pour être les équivalents américains du prix Nobel de physiologie ou médecine, ont été attribués le 23 septembre de cette même année. Ils distinguent six chercheurs : en recherche médicale fondamentale, Demis Hassabis et John Jumper pour l’élaboration d’AlphaFold, une méthode d’intelligence artificielle (IA) de détermination rapide de la structure tridimensionnelle des protéines ; en recherche clinique, David Huang, James Fujimoto et Eric Swanson pour la mise au point d’un dispositif permettant l’appréciation rapide de l’état physique de la rétine, l’OCT (pour optical coherence tomography) ; enfin, Piet Borst, pour l’ensemble de sa carrière de chercheur et d’enseignant.
Le prix Albert-Lasker pour la recherche médicale fondamentale
Le prix Albert-Lasker pour la recherche fondamentale a été attribué au Britannique Demis Hassabis et à l’Américain John Jumper pour l’élaboration du logiciel d'intelligence artificielle AlphaFold.
Les protéines sont constituées de l’enchaînement d’acides aminés. Chacune d’entre elles se replie sur elle-même en une configuration tridimensionnelle caractéristique. Cette structure détermine leur fonction biologique, essentiellement liée à des interactions avec d’autres molécules. La compréhension de ces interactions au niveau atomique est nécessaire à celle des processus biologiques. Depuis le milieu du xxe siècle, différentes techniques ont été utilisées pour déterminer les structures protéiques : diffraction des rayons X, résonance magnétique nucléaire, spectroscopies diverses, rayonnement synchrotron, cryoélectromicroscopie, etc. Leur mise en œuvre s’étant révélée bien souvent lente et coûteuse, peu de protéines ont finalement vu leur structure déterminée au niveau atomique. On connaît pourtant la structure de chaque acide aminé, ses capacités de liaisons chimiques, les angles de ces liaisons entre acides aminés et les possibilités d’échanges de liaisons faibles en énergie. Il s’avère donc théoriquement possible de calculer une structure probable à partir de la séquence en acides aminés d’une protéine. Les travaux en ce sens remontent aux années 1980, et ne se sont pas révélés très efficaces, jusqu’à ce que ces méthodes de calcul soient bouleversées à la fin des années 2000 par les techniques d’apprentissage profond (deep learning) appliquées au calcul de structures des protéines.
Demis Hassabis et John Jumper, tous deux du Google DeepMind, de Londres, ont mis au point un algorithme de prédiction des structures protéiques à partir des séquences en acides aminés. En 2018, des techniques d’apprentissage machine (machine learning) sont introduites dans les travaux portant sur ce sujet. Le machine learning laisse les algorithmes découvrir des motifs récurrents, ici dans les séquences de protéine, et apprend à les rechercher dans d’autres situations. Dans le cas des protéines, les motifs d’apprentissage – ainsi certaines structures récurrentes dans des protéines associées à des séquences d’acides aminés – ont été accumulés au cours des études antérieures. Cette première étape a constitué un changement décisif dans l’approche du calcul de structures de protéines. Le « coup de génie » de Demis Hassabis et John Jumper a été d’introduire dans les motifs d’apprentissage non seulement les données structurales déjà connues, mais aussi des données fonctionnelles – sites de phosphorylation, sites d’interactions entre domaines de protéines, séquences de régions transmembranaires, etc. – ou évolutives – comme des motifs conservés dans des familles de protéines et (ou) au cours de l’évolution. Leur logiciel, appelé AlphaFold, réalisé en collaboration avec l’European Molecular Biology Laboratory-European Bioinformatics Institute (EMBL-EBI) et dont les principes[...]
La suite de cet article est accessible aux abonnés
- Des contenus variés, complets et fiables
- Accessible sur tous les écrans
- Pas de publicité
Déjà abonné ? Se connecter
Écrit par
- Gabriel GACHELIN : chercheur en histoire des sciences, université Paris VII-Denis-Diderot, ancien chef de service à l'Institut Pasteur
Classification
Médias
Voir aussi
- RÉTINE
- INTERFÉROMÉTRIE
- STRUCTURE, biologie
- BIOLOGIE MOLÉCULAIRE
- CHROMOSOMES
- OPHTALMOLOGIE
- HASSABIS DEMIS (1976- )
- TOMOGRAPHIE
- GÉNIE BIOMÉDICAL ET BIO-INGÉNIERIE
- PROTÉINES
- IMAGE ou REPRÉSENTATION TRIDIMENSIONNELLE (3D)
- LASKER PRIX
- JUMPER JOHN (1985- )
- FUJIMOTO JAMES G. (1957- )
- BORST PIET (1934- )
- HUANG DAVID
- SWANSON ERIC A.