- 1. L’ADN, molécule support de l’information génétique
- 2. Le séquençage « historique et classique » de l’ADN : la méthode de Sanger
- 3. Le séquençage NGS et ses approches à haut débit
- 4. La troisième génération de séquençage
- 5. Le single cell sequencing (SCS)
- 6. Ordonner les données de séquençage
- 7. Exploiter les données de NGS
- 8. Évolution des techniques et perspectives
- 9. Bibliographie
SÉQUENÇAGE HAUT DÉBIT DE L'ADN
Article modifié le
La génomique, qui regroupe tout un ensemble d’analyses parmi lesquelles le séquençage de l’ADN, est une discipline de la biologie moderne dédiée à l’étude des génomes. Elle permet, à l’échelle d’un organisme, d’étudier la structure de ses gènes, leurs variations, leur fonctionnement, le contrôle de leur expression ainsi que leur évolution. La variation d’expression génique s’étudie le plus souvent pour un tissu particulier voire de plus en plus pour une cellule unique. Le séquençage de l'ADN est ainsi devenu un outil précieux pour les études génomiques en recherche scientifique et médicale. En effet, connaître l’organisation des génomes et leur variabilité permet de caractériser les espèces, de comprendre les mécanismes conduisant à des mutations affectant les individus et nécessaires pour leur adaptation aux conditions du milieu, mais également d’identifier les gènes impliqués dans tel ou tel phénotype et dans certaines maladies afin de pouvoir les diagnostiquer et, peut-être, essayer de les prendre en charge.
Le séquençage de l’ADN apparaît à la fin des années 1970 ; à l’époque, cette technique fait déjà évoluer de façon remarquable la biologie moléculaire à travers, entre autres, l’arrivée progressive de systèmes d’analyse des séquences de plus en plus autonomes, surtout durant les années 1990, bien que ne traitant qu’un maximum de quelques séquences à la fois. Cependant, depuis les années 2000, on peut parler de « révolution technologique » dans le domaine de la génomique avec l’émergence de plusieurs technologies innovantes, y compris dans le domaine du séquençage, qui ont permis d’entrer réellement dans l’ère haut débit ou NGS (nextgenerationsequencing, « séquençage de nouvelle génération », de « deuxième génération » ou « haut débit »). Ces outils extrêmement automatisés et ultraperformants, puisque traitant jusqu’à plusieurs millions de séquences en parallèle, ouvrent ainsi de nouvelles perspectives majeures, ne serait-ce qu’en médecine et en agronomie. Depuis les années 2010, les approches NGS ont elles-mêmes encore grandement évolué et permettent désormais de séquencer directement la molécule d’ADN ; on parle alors de NNGS (nextnextgenerationsequencing, ou « séquençage de troisième génération »).
Ces avancées exceptionnelles ont également eu comme impact un effondrement vertigineux des coûts financiers pour l’accès à l’information génomique. Ainsi, la première version du génome humain réalisée à l’aide de multiples séquenceurs de première génération a été publiée après plus de treize ans de travail et grâce à l’investissement de vingt laboratoires à travers le monde, pour un coût avoisinant les trois milliards de dollars. Cette information est d’ailleurs encore précieuse aujourd’hui pour l’étude des maladies génétiques rares. À peine vingt années plus tard, il est possible de faire séquencer presque entièrement son propre génome en un jour et pour seulement un millier de dollars !
Après un bref rappel sur l’essor historique du séquençage, une présentation plus détaillée décrira les évolutions qui ont mené cette technologie vers le haut débit et la place d’importance que le NGS occupe aujourd’hui dans les laboratoires, offrant ainsi un outil de haut potentiel dans la réalisation de nombreuses analyses scientifiques.
L’ADN, molécule support de l’information génétique
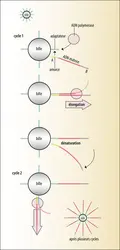
L’acide désoxyribonucléique (ADN) est présent dans le noyau de quasiment toutes les cellules vivantes, soit sous une forme très compactée, les chromosomes, soit relâchée. Il prend alors la forme d’une longue chaîne en double hélice composée de deux brins antiparallèles et complémentaires (le brin sens dit 5’-3’ et l’antisens 3’-5’), reliés l’un à l’autre par des liaisons hydrogène entre désoxyribonucléotides (dNTP). Ceux-ci[...]
La suite de cet article est accessible aux abonnés
- Des contenus variés, complets et fiables
- Accessible sur tous les écrans
- Pas de publicité
Déjà abonné ? Se connecter
Écrit par
- Véronique BLANQUET : professeure de génétique, université de Limoges
- Nathalie DUPRAT : ingénieure d'études en techniques biologiques
- Lionel FORESTIER : ingénieur d'études en expérimentation et techniques biologiques
Classification
Médias
Voir aussi
- AMORCEUR DE POLYMÉRISATION
- HÉRÉDITAIRES MALADIES ou MALADIES GÉNÉTIQUES
- NUCLÉOTIDES
- ADN POLYMÉRASE
- ARN MESSAGER ou ARNm
- CYTOSINE
- ADÉNINE
- RÉPLICATION, biologie moléculaire
- SCIENCES HISTOIRE DES, XXe XXIe s.
- SÉQUENÇAGE, génétique moléculaire
- GÉNOME
- ARN NON CODANTS ou ARNnc
- BIOLOGIE MOLÉCULAIRE
- INFORMATION GÉNÉTIQUE
- EXPRESSION GÉNÉTIQUE
- EXON
- GÉNÉTIQUE MOLÉCULAIRE
- MOLÉCULES BIOLOGIQUES, structure et fonction
- BIOLOGIE HISTOIRE DE LA
- MICROBIOTES
- MÉTAGÉNOMIQUE