- 1. L’ADN, molécule support de l’information génétique
- 2. Le séquençage « historique et classique » de l’ADN : la méthode de Sanger
- 3. Le séquençage NGS et ses approches à haut débit
- 4. La troisième génération de séquençage
- 5. Le single cell sequencing (SCS)
- 6. Ordonner les données de séquençage
- 7. Exploiter les données de NGS
- 8. Évolution des techniques et perspectives
- 9. Bibliographie
SÉQUENÇAGE HAUT DÉBIT DE L'ADN
Le séquençage « historique et classique » de l’ADN : la méthode de Sanger
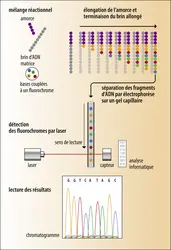
Le séquençage consiste donc à déterminer l'ordre dans lequel se succèdent, de l’extrémité 5’ à l’extrémité 3’, les quatre types de nucléotides (A, C, G, T) qui composent une molécule d’ADN. Après quelques différentes réalisations sporadiques (premiers résultats en 1963 fondés sur une chimie complexe), le séquençage dit de première génération – encore utilisé aujourd’hui – et développé sur la méthode de Frederick Sanger – son inventeur en 1977 qui reçoit pour cela son second prix Nobel en 1980 – s’affirme à la findes années 1970, presque vingt ans après la découverte de la structure de l'ADN. Il repose sur le mécanisme de réplication de l’ADN, au cours duquel la matrice ADN sous forme simple brin est recopiée par une enzyme, l’ADN polymérase, qui allonge un brin complémentaire à partir d’un petit fragment, l’amorce, en utilisant les quatre différents dNTPs présents dans le milieu. Une amorce (primer en anglais) est une courte séquence d’ADN synthétisée chimiquement qui se fixe exactement sur une séquence complémentaire (hybridation) de l’ADN simple brin, reconstituant localement un court ADN double brin à partir duquel toutes sortes d’opérations, telles que la copie ou l’amplification, sont réalisables. Lors d’une réaction de séquençage, les quatre dNTP sont donc ajoutés, ainsi qu’une faible proportion de didésoxyribonucléotides (ddNTP, qui diffèrent des dNTP par l’absence d’un groupe OH sur leur ribose, empêchant l’ajout d’un nucléotide suivant lors de l’élongation). Chacun des quatre ddNTP est couplé à un marqueur fluorescent distinct, rendant possible leur lecture multiplexe lors de la réalisation des séquences. Lorsqu’un ddNTP s’incorpore, il empêche la fixation du nucléotide suivant, stoppant ainsi l’élongation. Il résulte de cette dernière un mélange de fragments d’ADN de tailles croissantes, selon l’endroit où le ddNTP s’est incorporé. Si l’on sépare ces fragments marqués dans un champ électrique (électrophorèse), l’échelonnement des marqueurs indique la séquence. Le développement des séquenceurs de première génération – sociétés Applied BiosystemsTM (Life TechnologiesTM à présent), Roche, Beckman Coulter – a permis de prendre en charge, de façon de plus en plus autonome la séparation par électrophorèse et l’identification des différents fragments marqués. Les signaux spécifiques de chaque ddNTP sont ainsi détectés lors de leur passage devant un laser d’excitation puis traduits sous forme de chromatogrammes (ou électrophorégrammes). La séquence du fragment se lit directement sur ce dernier. Dans ce type de séquenceurs automatiques, l’électrophorèse se déroule systématiquement dans des capillaires, dont le nombre peut aller jusqu’à 384, augmentant ainsi sensiblement le débit.
Toutefois, même si quelques dizaines voire centaines d’échantillons peuvent être analysés simultanément, on ne parle pas encore de haut débit : d’une part, la méthode nécessite diverses étapes en amont dont certaines entièrement manuelles ; d’autre part, elle est limitée à l’obtention, en une heure environ, de séquences à peine supérieures à un millier de nucléotides consécutifs par capillaire. Les échelles de grandeur des séquences déterminées sont donc de l’ordre de centaines voire de quelques milliers pour le nombre de fragments traités, et la quantité de données gérées en aval se compte en kilobases (kb) pour atteindre au maximum quelques mégabases (Mb). Cette technique a fait évoluer de façon remarquable la génétique moléculaire à travers, entre autres, l’arrivée progressive de systèmes d’analyse informatique de plus en plus autonomes, surtout durant les années 1990, bien que ne traitant qu’un maximum de quelques séquences à la fois. Une des réussites les[...]
La suite de cet article est accessible aux abonnés
- Des contenus variés, complets et fiables
- Accessible sur tous les écrans
- Pas de publicité
Déjà abonné ? Se connecter
Écrit par
- Véronique BLANQUET : professeure de génétique, université de Limoges
- Nathalie DUPRAT : ingénieure d'études en techniques biologiques
- Lionel FORESTIER : ingénieur d'études en expérimentation et techniques biologiques
Classification
Médias