- 1. L’ADN, molécule support de l’information génétique
- 2. Le séquençage « historique et classique » de l’ADN : la méthode de Sanger
- 3. Le séquençage NGS et ses approches à haut débit
- 4. La troisième génération de séquençage
- 5. Le single cell sequencing (SCS)
- 6. Ordonner les données de séquençage
- 7. Exploiter les données de NGS
- 8. Évolution des techniques et perspectives
- 9. Bibliographie
SÉQUENÇAGE HAUT DÉBIT DE L'ADN
Le séquençage NGS et ses approches à haut débit
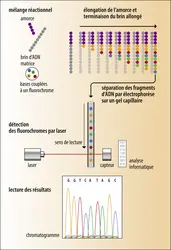
Le séquençage de deuxième génération apparaît de façon concrète en 2005, en réponse à un besoin de plus en plus important des laboratoires pour séquencer rapidement et à faibles coûts. Plusieurs approches totalement novatrices sont alors développées, les deux principales, toujours d’actualité en 2020, étant commercialisées par les sociétés Illumina® et Life TechnologiesTM. Globalement, ces techniques fonctionnent en trois étapes successives : la préparation et l'amplification de molécules d'ADN à analyser (création de librairies, mot « franglais » utilisé pour « bibliothèques ») ; l'incorporation et la détection en temps réel des bases complémentaires du brin à séquencer et l’analyse bio-informatique permettant la lecture de la totalité ou presque de la séquence matrice initiale ; son exploitation suivant l’application et les objectifs visés.
La préparation des librairies de fragments d’ADN
La première étape, commune aux deux technologies, consiste à préparer des collections – les librairies – de courts fragments, en général de taille définie, issus aléatoirement de l’ensemble du génome, ou d’une partie plus ciblée de l’échantillon à séquencer. Pour ce faire, l’ADN génomique ou complémentaire (synthétisé à partir d’ARN par transcription inverse lors des études transcriptomiques) est fragmenté par voie enzymatique ou mécanique. De courts fragments d’ADN appelés adaptateurs, spécifiques à chaque technologie, vont ensuite être attachés aux extrémités des fragments afin de servir d’amorces pour la suite les étapes de fixation et d’amplification, puis de séquençage. Ces adaptateurs peuvent en outre comporter une séquence particulière, un « tag », qui va permettre d’identifier tous les fragments provenant d’un même échantillon. Ces « index » ou « code-barres » (suivant la technologie employée), identifiés lors de l’étape de séquençage permettent de mélanger plusieurs échantillons différents pour utiliser pleinement les capacités de chaque système de séquençage.
L’amplification « clonale »
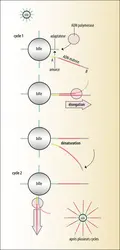
Chacun des fragments contenus dans les librairies doit être ensuite multiplié – amplifié – à l’identique, individuellement et en proportions équivalentes. La première étape va donc consister à isoler physiquement chaque fragment d’ADN dans le mélange en fixant chacun soit sur un support unique (bille) soit sur une zone dédiée d’un support commun (flowcell). Puis l’amplification de ces fragments fixés (chacun équivalent à un clone) est réalisée par PCR (polymerasechainreaction). Cette technique permet de recopier rapidement une séquence précise d’ADN et de doubler le nombre de copies de cet ADN à chaque cycle de l’amplification. Suivant la technologie NGS employée, la PCR sera réalisée en émulsion ou « en pont ».
Au cours de la PCR en émulsion (Life TechnologiesTM), les fragments sont dilués et fixés sur des microbilles via les adaptateurs, de sorte que, le plus souvent, une seule molécule d’ADN se fixe sur chaque bille (fixation monoclonale). Les billes sont ensuite recouvertes d'un film liquide aqueux contenant tous les réactifs nécessaires à l’amplification par PCR et mélangées dans un tube rempli d'huile. Chaque bille est ainsi isolée des autres dans son propre microréacteur et toutes les billes vont subir en même temps les différentes étapes d’amplification. Chaque fragment va ainsi être amplifié à la surface de chaque bille.
Dans le cas de la PCR en pont (Illumina®), l’ensemble des fragments à cloner est dilué et réparti à la surface d'une plaque en verre (dite flowcell), de façon à ce que les molécules soient éloignées les unes des autres. Celles-ci vont s’hybrider sur des amorces universelles dont la plaque est recouverte, par l’intermédiaire[...]
La suite de cet article est accessible aux abonnés
- Des contenus variés, complets et fiables
- Accessible sur tous les écrans
- Pas de publicité
Déjà abonné ? Se connecter
Écrit par
- Véronique BLANQUET : professeure de génétique, université de Limoges
- Nathalie DUPRAT : ingénieure d'études en techniques biologiques
- Lionel FORESTIER : ingénieur d'études en expérimentation et techniques biologiques
Classification
Médias